I ricercatori della Drexel University hanno individuato un metodo per identificare rapidamente ed etichettare le versioni mutate del virus che causa la COVID-19. La loro analisi preliminare, utilizzando le informazioni di un database globale di informazioni genetiche raccolte dai test del coronavirus, suggerisce che ci siano almeno da 6 a 10 versioni leggermente diverse del virus che infetta le persone in America, alcune delle quali sono uguali o sono evoluzioni successive di ceppi provenienti direttamente dall’Asia, mentre altri sono uguali a quelli trovati in Europa.
Lo strumento di analisi genetica può identificare se un virus è cambiato geneticamente. È quindi possibile utilizzare il modello per classificare i virus con piccole differenze genetiche utilizzando tag chiamati Informative Subtype Markers (ISM).
Applicando lo stesso metodo per elaborare i dati genetici virali è possibile rilevare e classificare rapidamente lievi variazioni genetiche nel SARS-CoV-2, il nuovo coronavirus che causa la COVID-19, secondo quanto riporta il gruppo di studio in un articolo recentemente pubblicato sull’archivio preliminare di ricerca, bioRxiv. Lo strumento di analisi genetica che genera queste etichette è pubblicamente disponibile per i ricercatori COVID-19 su GitHub.
“I tipi di virus SARS-CoV-2 che vediamo nei test dall’Asia e dall’Europa sono diversi dai tipi che vediamo in America“, ha dichiarato Gail Rosen, Ph.D., professore al Drexel College of Engineering, che ha guidato lo sviluppo dello strumento. “L’identificazione delle variazioni ci consente di vedere come il virus è cambiato mentre viaggiava da una popolazione all’altra. Può anche mostrarci le aree in cui il distanziamento sociale ha avuto successo nell’isolare COVID-19“.
Lo strumento ISM, sviluppato da Rosen e da un team focalizzato, è particolarmente utile perché non richiede l’analisi dell’intera sequenza genetica del virus per identificare le sue mutazioni. Nel caso del SARS-CoV-2, ciò significa ridurre il codice genetico del virus lungo 30.000 basi a un’etichetta di sottotipo lunga 17 basi.
“È l’equivalente della scansione di un codice a barre invece di digitare il numero di codice completo del prodotto“, ha affermato Rosen. “E questo ci mette in grado di passare all’analisi di livello superiore molto più velocemente. Ad esempio, un processo più veloce nello studio delle versioni dei virus potrebbe influire sugli esiti sanitari. Oppure, i funzionari della sanità pubblica possono verificare se i nuovi casi sono il risultato della trasmissione locale o provengono da altre regioni degli Stati Uniti o altre parti del mondo.
Mentre queste differenze genetiche potrebbero non essere sufficienti per delineare un nuovo ceppo di virus, il gruppo di Rosen suggerisce di considerare questi “sottotipi” geneticamente significativi, dove vengono trovati e quanto sono prevalenti in queste aree, i dati sono abbastanza granulari da essere utili.
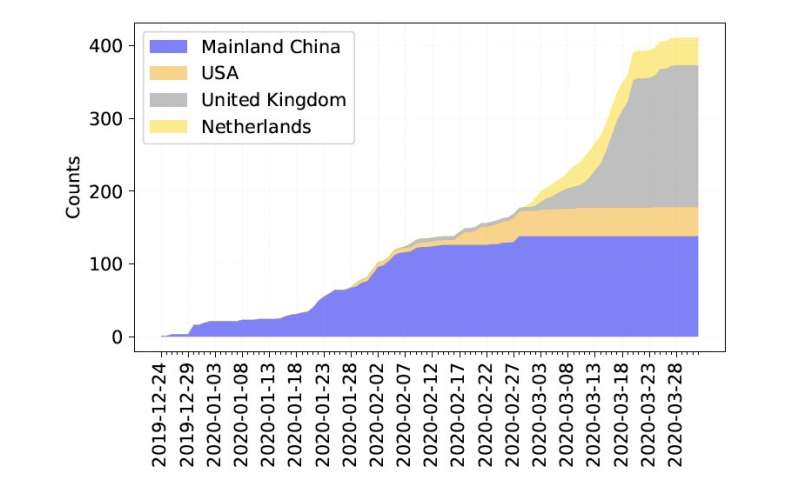
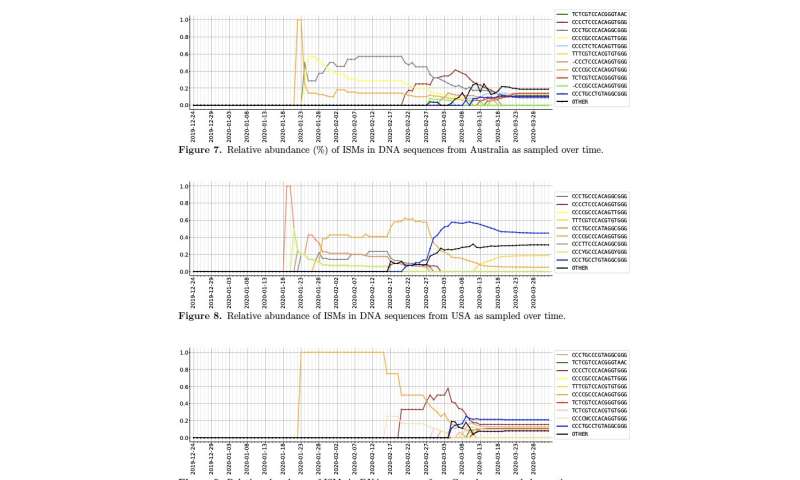
“Questo ci consente di vedere l’impronta digitale molto specifica della COVID-19 da ogni regione del mondo e di osservare da vicino le regioni più piccole per vedere come divergono“, ha detto Rosen. “La nostra analisi preliminare, utilizzando dati disponibili pubblicamente in tutto il mondo, sta dimostrando che la combinazione di sottotipi di virus rilevati a New York è molto simile a quella riscontrata in Austria, Francia ed Europa centrale, ma non in Italia. E il sottotipo proveniente dall’Asia, che è stato rilevato qui all’inizio della pandemia non si è diffuso molto, invece stiamo vedendo un nuovo sottotipo che esiste solo in America come quello più diffuso nello stato di Washington e sulla costa occidentale“.
Oltre ad aiutare gli scienziati a capire come il virus sta cambiando e si sta diffondendo, questo metodo può anche rivelare la parte del suo codice genetico che sembra rimanere resistente alle mutazioni, una scoperta che potrebbe essere sfruttata dai trattamenti per combattere il virus.
“Stiamo vedendo che le due parti del virus che sembrano non mutare sono quelle responsabili del suo ingresso in cellule sane e del confezionamento del suo RNA“, ha detto Rosen. “Entrambi sono obiettivi importanti per comprendere la risposta immunitaria del corpo, identificare terapie antivirali e progettare vaccini“.
Fonte: Phys.org